VCF (биоинформатика)
| Variant Call Format | |
|---|---|
 | |
| Расширение |
.vcf |
| Разработчик | 1000 Genomes Project |
| Последний выпуск | 4.5 (9 октября 2024) |
| Тип формата | Genomic sequence format |
| Расширен из | Tab-separated values |
| Развит в | gVCF |
VCF (от англ. Variant Call Format — формат вызова вариантов) — стандартный формат текстового файла, используемый в генетической биоинформатике для хранения вариаций последовательности генов (последовательности ДНК).
Разработан в 2010 году для проекта «1000 геномов» и с тех пор использовался в других крупномасштабных проектах по генотипированию и секвенированию ДНК[1][2]. Получил распространение благодаря своей относительной простоте и масштабируемости[3][4]. Имеется множество инструментов для редактирования и обработки VCF-файлов, включая VCFtools, выпущенный вместе с форматом VCF в 2011 году, и BCFtools, который был включен в состав SAMtools до тех пор, пока не был выделен в независимый пакет в 2014 году[1][5].
В настоящее время[уточнить] действует стандарт версии 4.5[6][7], а проект «1000 геномов» разработал собственную спецификацию[8].
На основе VCF были разработаны дополнительные форматы файлов, среди них — геномный VCF (gVCF) — расширенный формат, который включает дополнительную информацию о «блоках»[9][10].

Заголовок VCF
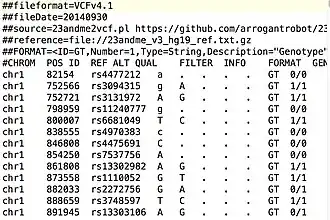
Заголовок начинает файл и содержит метаданные, описывающие тело файла. Строки заголовка обозначаются как начинающиеся с# . Специальные ключевые слова в заголовке обозначены знаком ## . Рекомендуемые ключевые слова включаютfileformat ,fileDate иreference .
Заголовок содержит ключевые слова, которые описывают поля.
Столбцы VCF
В VCF файлах за заголовком и разделено табуляцией на 8 обязательных столбцов и дополнительные столбцы, которые могут использоваться для записи другой информации об образце(ах).
| Имя | Краткое описание | |
|---|---|---|
| 1 | CHROM | Название последовательности (обычно хромосомы), в которой вызывается вариация. Эту последовательность обычно называют «референтной последовательностью», то есть последовательностью, относительно которой варьируется данный образец. |
| 2 | POS | Позиция вариации в данной последовательности, начиная с 1. |
| 3 | ID | Идентификатор вариации, например, идентификатор dbSNP rs или, если неизвестно, «.». Несколько идентификаторов должны быть разделены точкой с запятой без пробелов. |
| 4 | REF | Референтная база (или базы в случае инделя) в заданной позиции в заданной референтной последовательности. |
| 5 | ALT | Список альтернативных аллелей в этой позиции. |
| 6 | QUAL | Показатель качества, связанный с выводом данных аллелей. |
| 7 | FILTER | Флаг, указывающий, какой из заданного набора фильтров вариация не прошла или ПРОШЛА, если все фильтры были пройдены успешно. |
| 8 | INFO | Расширяемый список пар ключ-значение (полей), описывающих вариацию. Ниже приведены некоторые общие поля. Несколько полей разделяются точкой с запятой с необязательными значениями в формате: < key > = < data > [,data] . |
| 9 | FORMAT | (Необязательный) расширяемый список полей для описания образцов. Ниже приведены некоторые общие поля. |
| + | SAMPLE | Для каждого (необязательного) образца, описанного в файле, указаны значения полей, перечисленных в FORMAT |
Общие поля INFO
Разрешены произвольные ключи, хотя следующие подполя зарезервированы (хотя и необязательны):[11]
| Имя | Краткое описание |
|---|---|
| АА | предковый аллель |
| АС | количество аллелей в генотипах, для каждого аллеля ALT, в том же порядке, как указано |
| AF | Частота аллелей для каждого аллеля ALT в том же порядке, как указано (используйте это при оценке на основе первичных данных, а не генотипов) |
| AN | общее число аллелей в названных генотипах |
| BQ | Базовое качество RMS в этой позиции |
| CIGAR | сигарная нить, описывающая, как выровнять альтернативный аллель с референтным аллелем |
| DB | членство в dbSNP |
| DP | объединенная глубина по всем образцам, например DP=154 |
| END | конечная позиция варианта, описанного в этой записи (для использования с символическими аллелями) |
| Н2 | членство в hapmap2 |
| Н3 | членство в hapmap3 |
| MQ | Качество отображения RMS, например MQ=52 |
| МQ0 | Количество чтений MAPQ == 0, охватывающих эту запись |
| NS | Количество образцов с данными |
| SB | смещение нити в этой позиции |
| SOMATIC | указывает на то, что запись является соматической мутацией, для геномики рака |
| VALIDATED | подтверждено последующим экспериментом |
| 1000G | членство в 1000 Геномов |
Все остальные поля информации определяются в заголовке .vcf.
Общие поля FORMAT
| Имя | Краткое описание |
|---|---|
| AD | Глубина чтения для каждого аллеля |
| ADF | Глубина считывания для каждого аллеля на прямой цепи |
| ADR | Глубина считывания для каждого аллеля на обратной цепи |
| DP | Глубина чтения |
| EC | Ожидаемое количество альтернативных аллелей |
| FT | Фильтр, показывающий, был ли этот генотип «вызван» |
| GL | Вероятности генотипа |
| GP | Апостериорные вероятности генотипа |
| GQ | Условное качество генотипа |
| GT | Генотип |
| HQ | Качество гаплотипа |
| MQ | Качество отображения RMS |
| PL | Вероятности генотипа по шкале Phred, округленные до ближайшего целого числа |
| PQ | Качество фазировки |
| PS | Фазовый набор |
Все остальные поля формата определяются в заголовке .vcf.
См. также
- Формат FASTA, используемый для представления последовательностей генома.
- Формат FASTQ, используемый для представления результатов секвенирования ДНК вместе с показателями качества.
- Формат SAM, используемый для представления результатов секвенирования генома, сопоставленных с последовательностями генома.
- Формат GVF (Genome Variation Format) — расширение на основе формата GFF3 .
- Глобальный альянс по геномике и здоровью (GA4GH), группа, возглавляющая управление и расширение формата VCF.[12] Спецификация VCF больше не поддерживается проектом 1000 Genomes.[13]
- Геном человека
- Полиморфизм одиночных нуклеотидов (SNP)
Примечания
- 1 2 Danecek, Petr; Auton, Adam; Abecasis, Goncalo; Albers, Cornelis A.; Banks, Eric; DePristo, Mark A.; Handsaker, Robert E.; Lunter, Gerton; Marth, Gabor T.; Sherry, Stephen T.; McVean, Gilean; Durbin, Richard (1 августа 2011). The variant call format and VCFtools. Bioinformatics. 27 (15): 2156–2158. doi:10.1093/bioinformatics/btr330. ISSN 1367-4803. PMC 3137218. PMID 21653522.
- ↑ Ossola, Alexandra (2015-03-20). The Race to Build a Search Engine for Your DNA. IEEE Spectrum. Дата обращения: 2015-03-22.
- ↑ Understanding VCF format | Human genetic variation (англ.). EMBL-EBI. Дата обращения: 10 ноября 2023. Архивировано 20 апреля 2023 года.
- ↑ Garrison, Erik; Kronenberg, Zev N.; Dawson, Eric T.; Pedersen, Brent S.; Prins, Pjotr (31 мая 2022). A spectrum of free software tools for processing the VCF variant call format: vcflib, bio-vcf, cyvcf2, hts-nim and slivar. PLOS Computational Biology. 18 (5): e1009123. Bibcode:2022PLSCB..18E9123G. doi:10.1371/journal.pcbi.1009123. ISSN 1553-734X. PMC 9286226. PMID 35639788.
- ↑ Danecek, Petr; Bonfield, James K; Liddle, Jennifer; Marshall, John; Ohan, Valeriu; Pollard, Martin O; Whitwham, Andrew; Keane, Thomas; McCarthy, Shane A; Davies, Robert M; Li, Heng (29 января 2021). Twelve years of SAMtools and BCFtools. GigaScience. 10 (2). doi:10.1093/gigascience/giab008. ISSN 2047-217X. PMC 7931819. PMID 33590861.
- ↑ [https://samtools.github.io/hts-specs/VCFv4.5.pdf VCF Specification 2024
- ↑ Specifications of SAM/BAM and related high-throughput sequencing file formats. GitHub. Дата обращения: 24 июня 2014.
- ↑ Encoding Structural Variants in VCF (Variant Call Format) version 4.0 | 1000 Genomes. Дата обращения: 20 октября 2016.
- ↑ GVCF - Genomic Variant Call Format. GATK. Broad Institute.
- ↑ gVCF Files. Illumina, Inc.. Дата обращения: 10 ноября 2023.
- ↑ VCF Specification. Дата обращения: 30 июля 2024.«VCF Specification» (PDF). Retrieved 30 July 2024.
- ↑ HTS format specifications. samtools.github.io. Дата обращения: 22 февраля 2022.
- ↑ Bio-IT World (амер. англ.). www.bio-itworld.com. Дата обращения: 26 октября 2018.